9 Minuten
Forschende haben eines der bislang detailliertesten virtuellen Gehirne erstellt: eine vollständige Simulation der Mauscortex, die einzelne Neuronen und ihre Verbindungen nachbildet. Dieses Modell ermöglicht neue Experimente zu Hirnwellen, Anfällen und neurologischen Erkrankungen, ohne invasive Eingriffe an lebenden Tieren durchzuführen.
How the virtual mouse cortex was built
Ein gemeinsames Team des Allen Institute (USA) und der University of Electro-Communications (Japan) nutzte umfangreiche Zell‑ und Gewebedatenbanken, anatomische Karten sowie neue, auf Effizienz optimierte Software, um ein dreidimensionales Modell des gesamten Mauscortex zusammenzustellen. Die virtuelle Struktur umfasst etwa 9 Millionen Neuronen, die über rund 26 Milliarden Synapsen in 86 miteinander verknüpften Regionen verbunden sind. Das System läuft auf Japans Supercomputer Fugaku und führt Berechnungen in der Größenordnung von Billiarden Operationen pro Sekunde durch, um realistische neuronale Aktivität zu simulieren.
Zum Vergleich: Ein echtes Mausgehirn enthält ungefähr 70 Millionen Neuronen und ist etwa so groß wie eine Mandel. Die simulierte Cortex‑Region bildet daher nur einen Teil des gesamten Organs ab, bewahrt aber entscheidende strukturelle und dynamische Details, die biologische Schaltkreise nachbilden. Die Arbeit demonstriert, dass großskalige, biologisch fundierte Gehirnmodelle mit ausreichend Rechenressourcen und präzisen biologischen Daten realisierbar sind. Für die Modellierung nutzten die Forscher Zelltyp‑Atlanten, detaillierte Konnektomdaten und elektrophysiologische Messreihen als Referenzpunkte, um sowohl anatomische als auch funktionelle Aspekte abzubilden.
Technisch gesehen kombiniert die Simulation mehrere Ebenen: morphologische Modelle einzelner Neuronen, synaptische Verbindungsstatistiken, Schichtarchitektur der Cortexregionen und zeitliche Aktivitätsmuster. Parameter wie synaptische Verzögerungen, Leitungsgeschwindigkeiten und kurzfristige Plastizität wurden anhand experimenteller Daten kalibriert. Diese Mehrskaligkeit — von subzellulären Eigenschaften bis hin zu netzwerkweiten Oszillationen — macht das Modell besonders wertvoll für Untersuchungen, die sowohl molekulare als auch systemische Mechanismen betreffen.
What the simulation lets scientists do
Das Modell bietet Forschenden eine dynamische, 3D‑Karte, die zeigt, wie einzelne Neuronen feuern und sich im Zeitverlauf zu Netzwerken formieren. Diese Granularität ermöglicht es, Hypothesen zur Kognition, zur Ausbreitung epileptischer Anfälle, zur interhemisphärischen Kommunikation und zur Erzeugung von Hirnwellen in silico zu testen — bevor oder statt zeit- und kostenintensiver sowie invasiver Tierversuche.
.avif)
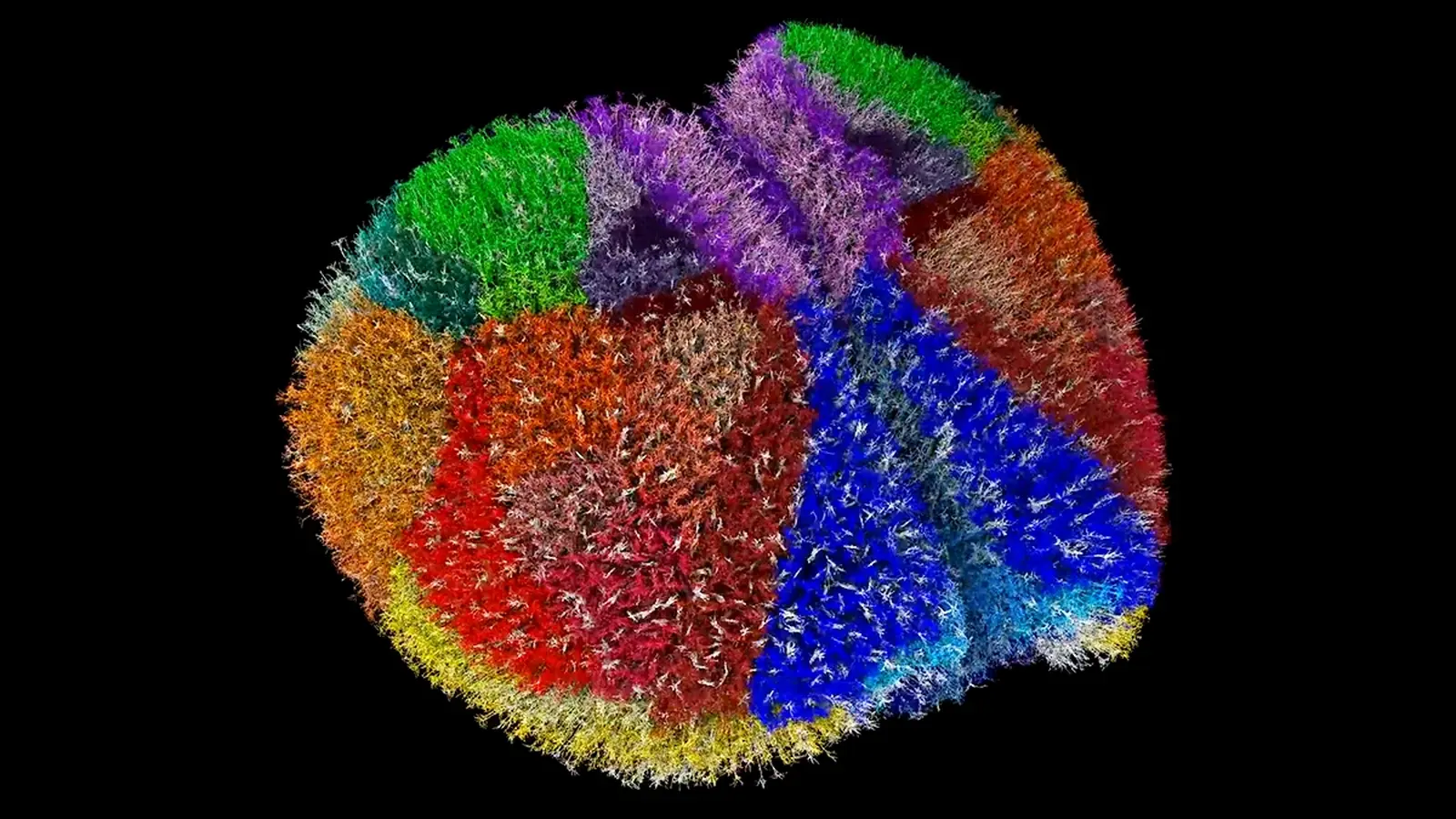
Die Simulation ermöglicht es Forschenden, die Aktivität einzelner Neuronen nachzuverfolgen.
Beispielsweise können Teams einen lokalisierten Aktivitätsausbruch im virtuellen Cortex auslösen, um zu beobachten, wie pathologische Rhythmen sich ausbreiten, oder untersuchen, wie synchronisierte Schwingungen zwischen beiden Hemisphären Aufmerksamkeits‑ähnliche Netzwerkzustände beeinflussen. Da die Software darauf abgestimmt ist, unnötige Berechnungen zu minimieren, werden lang andauernde Versuche und umfangreiche Parameterstudien auf aktuellen Supercomputern praktikabel.
Die Simulation bietet auch Werkzeuge zur Kontrolle und Messung: Stimulationsprotokolle können präzise positioniert werden, einzelne Synapsen können in ihrer Wirkstärke moduliert werden, und Topologien von Netzwerken lassen sich systematisch variieren. Solche in silico‑Experimente erlauben es, kausale Zusammenhänge zwischen zellulären Veränderungen und netzwerkweiten Effekten zu identifizieren — ein zentraler Aspekt für die Erforschung von Mechanismen neurologischer Erkrankungen und der Entwicklung zielgerichteter Interventionen.
„This shows the door is open,“ sagt der computational neuroscientist Anton Arkhipov vom Allen Institute. „We can run these kinds of brain simulations effectively with enough computing power. It's a technical milestone giving us confidence that much larger models are not only possible, but achievable with precision and scale.“ Diese Aussage betont: Mit wachsender Rechenleistung und verbesserten biologischen Daten wächst das Potenzial für noch umfassendere Modelle, die zunehmend realitätsnahe Vorhersagen erlauben.
Scientific context and implications
Großskalige Gehirnsimulationen stehen an der Schnittstelle von Neurowissenschaften, Hochleistungsrechnen und Data Science. Sie stützen sich auf detaillierte Zelltyp‑Atlanten, Konnektomkarten (die aufzeigen, wie Neuronen verdrahtet sind) und elektrophysiologische Messungen, die die Aktivität des Modells einschränken. Sind diese Eingabedaten präzise, können simulierte Netzwerke messbare Eigenschaften von echtem Gewebe reproduzieren — etwa Feuerungsraten, Synchronisationsmuster und Ausbreitungsgeschwindigkeiten von Wellen.
Aus klinischer Perspektive beschleunigen solche Modelle die Forschung zu neurodegenerativen Erkrankungen wie Alzheimer, weil sie eine systematische Untersuchung erlauben, wie zelluläre Veränderungen die Netzwerkdynamik über Zeiträume beeinflussen. Simulationen helfen zudem, die Anzahl lebender Tierversuche zu reduzieren, indem sie vielversprechende Interventionen vorselektieren und mögliche Nebenwirkungen auf die Netzwerkfunktion vorhersagen.
Wichtig ist, dass Modelle nicht nur qualitativ beschreiben, sondern quantitative Vorhersagen liefern — etwa Prognosen über Änderungen der Signalübertragung in bestimmten Schichten oder Vorläufermuster für pathologische Synchronisationen. Solche Einsichten können experimentelle Hypothesen fokussieren und die Planung von in vivo‑Studien effizienter gestalten.
Tadashi Yamazaki, Informatiker an der University of Electro‑Communications, hebt Fugakus breitere Rolle hervor: „Fugaku wird in vielen Bereichen der computational science eingesetzt, etwa in Astronomie, Meteorologie und Wirkstoffforschung, und trägt zur Lösung zahlreicher gesellschaftlicher Probleme bei. In diesem Fall nutzten wir Fugaku für eine Simulation neuronaler Schaltkreise.“ Die Kombination aus leistungsfähiger Hardware und optimierter Softwarearchitektur ist ein wesentlicher Faktor dafür, dass Simulationen in der Größenordnung von Millionen Neuronen überhaupt praktikabel werden.
Darüber hinaus erlaubt die Plattform explorative Analysen von Störanfälligkeiten — etwa wie Läsionen, Proteinhäufungen oder verringerte synaptische Effektstärke lokale und globale Netzwerkzustände verändern. Solche Analysen sind für die Translation von pathophysiologischen Befunden in therapeutische Strategien von großer Bedeutung.
Future prospects and challenges
Das Team, das das Modell entwickelt hat, nutzt es bereits, um Hirnwellen‑Synchronisation und die Interaktionen zwischen den Cortex‑Hemisphere zu untersuchen. Die Forscher haben jedoch ehrgeizigere Pläne: langfristig sollen Ganzhirnmodelle entstehen, inklusive Simulationen in menschlichem Maßstab, die sämtliche verfügbaren biologischen Details integrieren. Dieses Ziel ist mit mehreren erheblichen Herausforderungen verbunden.
- Data completeness: Die menschlichen Gehirne sind deutlich komplexer als ein Mauscortex; die Beschaffung notwendiger hochauflösender Zelltyp‑ und Konnektivitätskarten ist ein langfristiges, interdisziplinäres Unterfangen. Insbesondere fein differenzierte Neuronentypen, regionale Transcriptom‑Profile und Synapsenspezifität sind noch nicht vollständig kartiert.
- Computational cost: Simulationen im menschlichen Maßstab erfordern um Größenordnungen mehr Rechenleistung und weiter optimierte Algorithmen, damit sie praktikabel bleiben. Effiziente Parallelisierung, Speicheroptimierung und spezialisierte Numerik sind gefragt, ebenso wie die Entwicklung hardwarenaher Softwarelösungen.
- Validation: Modelle müssen kontinuierlich gegen experimentelle Daten validiert werden, damit simulierte Dynamiken biologisch begründet sind und nicht Artefakte modellbedingter Annahmen widerspiegeln. Validationsstrategien umfassen Vergleiche mit EEG/MEG, in vitro‑Aufzeichnungen, bildgebenden Verfahren und Verhaltensdaten.
Trotz dieser Herausforderungen stellt der aktuelle Forschungsansatz einen überzeugenden Proof‑of‑Concept dar. Durch die Kombination präziser biologischer Datensätze mit führender Supercomputing‑Infrastruktur haben Forschende einen bedeutenden Schritt in Richtung einer neuen Klasse von Werkzeugen unternommen, mit denen sich Gehirnfunktion und -dysfunktion systematisch untersuchen lassen.
Weitere technische Hürden betreffen die Modellierung von Gliazellen, Neuromodulation und Stoffwechselprozessen, die für die langfristige Stabilität und Pathogenese wichtig sind. Ebenso wird die Integration multimodaler Daten — Genexpression, Proteomik, Konnektivität und elektrophysiologische Messreihen — entscheidend für die Aussagekraft zukünftiger Modelle sein. Interdisziplinäre Zusammenarbeit zwischen Neurowissenschaftlern, Informatikern, Physikern und Klinikerinnen ist daher unerlässlich.
Expert Insight
Dr. Maya Fernandez, Neurowissenschaftlerin und computational modeler in einem universitären Neurotechnologie‑Labor, kommentierte die Arbeit: „Large‑scale cortical models mark a turning point for translational neuroscience. They let us probe mechanism‑level questions—how cellular pathology scales to circuit dysfunction—without the confounds present in living systems. The critical next step is integrating multimodal human data so simulations can meaningfully inform clinical strategies.“
Übersetzt und kontextualisiert bedeutet das: Groß‑skalige Cortex‑Modelle markieren einen Wendepunkt für die translationale Neurowissenschaft. Sie erlauben Untersuchungen auf Mechanismenebene — etwa wie zelluläre Pathologien zu circuit‑weiten Funktionsstörungen eskalieren — ohne die Störeinflüsse lebender Systeme. Der nächste kritische Schritt besteht in der Integration multimodaler humaner Daten, damit Simulationen klinische Strategien sinnvoll unterstützen können.
Fernandez betont, dass neben biologischer Genauigkeit auch Transparenz der Modellannahmen und Reproduzierbarkeit der Simulationen für die Akzeptanz in der klinischen Forschung entscheidend sind. Offene Datenformate, standardisierte Benchmarks und öffentlich zugängliche Validationsdatensätze werden hierbei eine Schlüsselrolle spielen.
Outlook
Diese Simulation wurde auf der SC25 Supercomputing Conference vorgestellt und ist online verfügbar. Sie stellt einen bedeutenden Fortschritt in der biologisch fundierten neuronalen Modellierung dar und zeigt, wie Supercomputer wie Fugaku Entdeckungen von der Grundlagenforschung bis zu potenziellen klinischen Anwendungen beschleunigen können. Mit fortschreitender Verbesserung von Datensätzen und Algorithmen werden virtuelle Gehirnplattformen immer mächtigere Instrumente zum Verständnis von Denken, Krankheit und den emergenten Dynamiken neuronalen Gewebes.
Langfristig könnten solche Plattformen helfen, personalisierte Therapiesimulationen zu entwickeln, in denen patientenspezifische Daten (Genomik, Bildgebung, elektrophysiologische Profile) in ein individuelles Modell eingespeist werden. Dies würde die Erforschung maßgeschneiderter Interventionen — etwa Stimulationsprotokolle, pharmakologische Strategien oder kombinierte Behandlungsansätze — ermöglichen und die Translation vom Modell zur Klinik beschleunigen.
Zudem eröffnen die Modelle Chancen für die Grundlagenforschung: Forscher können Fragen zur Entstehung bewusster Zustände, zur Rolle spezifischer Schichten bei Informationsverarbeitung oder zur Koordination großräumiger Netzwerke über verschiedene Frequenzbänder hinweg adressieren. Dabei bleibt die enge Verzahnung von Simulation und Experiment zentral, um Modelle iterativ zu verbessern und ihre Vorhersagekraft systematisch zu stärken.
Abschließend ist zu betonen, dass die Entwicklung virtueller Gehirne nicht nur eine technische Aufgabe ist, sondern auch ethische und regulatorische Fragen aufwirft. Transparenz, Datenethik und die verantwortungsvolle Nutzung solcher Simulationen müssen von Beginn an Bestandteil des wissenschaftlichen Diskurses sein, um Nutzen zu maximieren und Risiken zu minimieren.
Quelle: sciencealert

Kommentar hinterlassen