6 Minuten
Uralte Mikroben in Mammut-Überresten erhalten
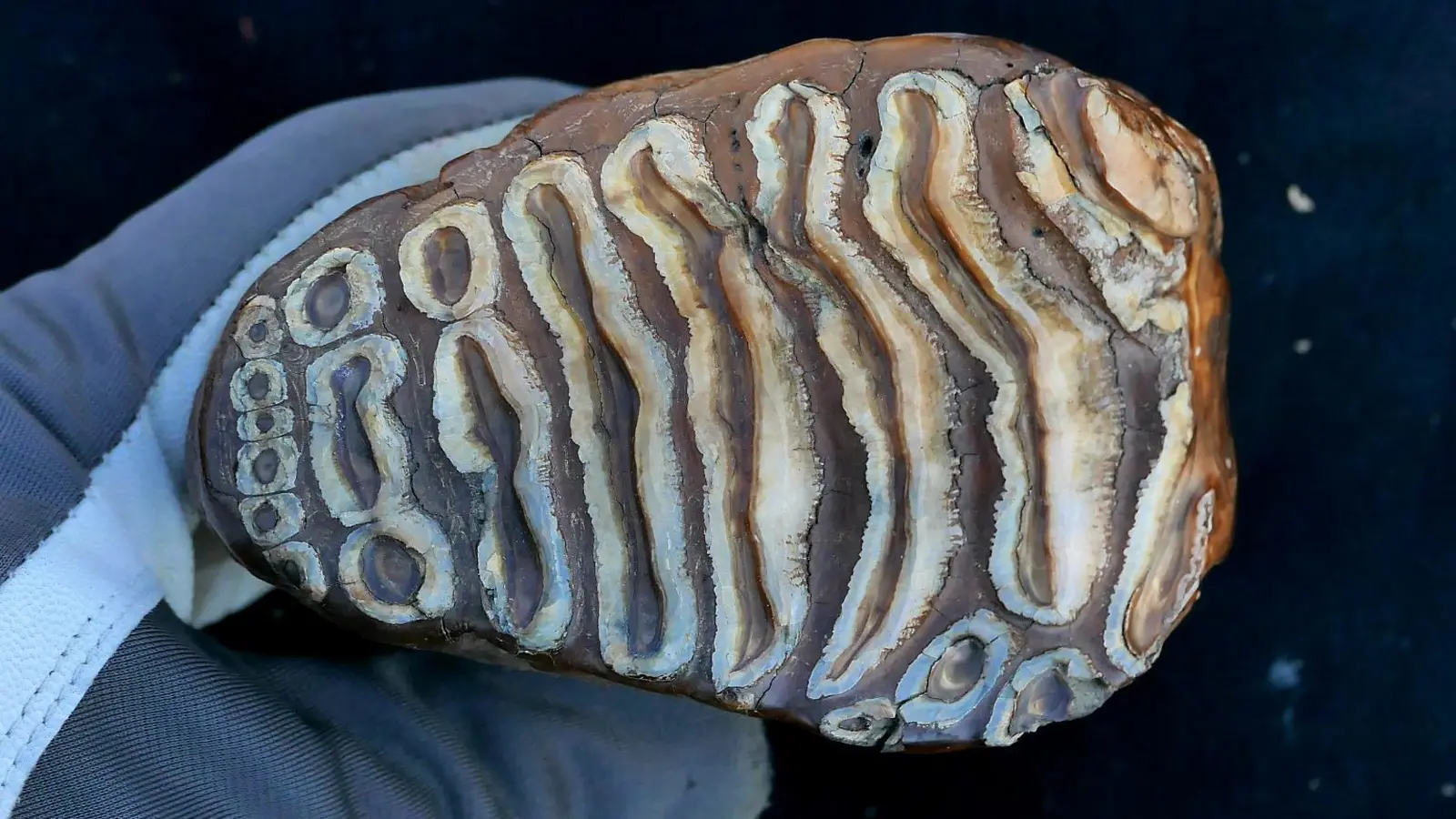
Ein internationales Forscherteam unter Leitung des Centre for Palaeogenetics hat mikrobielles DNA-Material aus Woll- und Steppemammut-Überresten gewonnen, das mehr als eine Million Jahre alt ist. Die Arbeit — veröffentlicht in Cell — zählt zu den ältesten je rekonstruierten, wirt-assoziierten mikrobiellen DNA-Proben und eröffnet ein neues Fenster auf die langfristigen Wechselwirkungen zwischen Pleistozän-Megafauna und ihren mikrobiellen Gemeinschaften. Die Forschenden untersuchten 483 Mammutproben, darunter 440, die zuvor noch nicht sequenziert worden waren, und nutzten moderne genomische und bioinformatische Methoden, um zwischen Mikroben zu unterscheiden, die mit den Mammuten lebten, und solchen, die das Material nach dem Tod kolonisierten.
Methods: authenticating microbial signals from deep time
Das Team kombinierte Hochdurchsatz-Sequenzierung, Analyse von Schadensmustern und strenge Kontaminationskontrollen, um antike, wirt-assoziierte Mikroben von modernen Umweltkontaminanten zu trennen. Zu den Proben gehörten Zähne, Stoßzähne und Knochenfragmente aus verschiedenen geografischen Regionen und Zeitabschnitten, darunter ein besonders bemerkenswertes Fundstück eines Steppemmutts, das auf etwa 1,1 Millionen Jahre datiert wurde. Die Laborarbeit folgte strikten Ancient-DNA-Protokollen: dedizierte Reinräume, negative Kontrollen und bioinformatische Filter, die die charakteristische Fragmentierung und Cytosin-zu-Thymin-Substitutionsmuster identifizieren, die für degradiertes antikes DNA-Material typisch sind.

Bioinformatische Verfahren halfen zudem, DNA-Sequenzen in Gruppen zu unterteilen, die wahrscheinlich aus dem lebenden Mikrobiom jedes Tieres stammen, und solche, die nach der Einbettung eingedrungen sind. Diese Unterscheidung basiert auf konsistenten taxonomischen Mustern zwischen Individuen, Erhaltungssignaturen und Vergleichen mit modernen Verwandten. Durch die Kombination dieser Evidenzlinien konnten die Forschenden partielle mikrobielle Genome rekonstruieren und nachweisen, dass bestimmte Linien über Hunderttausende von Jahren mit Mammuten koexistierten.
Key discoveries: persistent microbial lineages and potential pathogens
In dem Datensatz identifizierten die Autorinnen und Autoren sechs mikrobielle Kladen, die wiederholt mit Mammut-Überresten assoziiert waren. Dazu gehören Verwandte von Actinobacillus, Pasteurella, Streptococcus und Erysipelothrix — Gattungen, die heute sowohl harmlose Kommensalen als auch pathogene Stämme enthalten. Ein in der Studie gefundener Pasteurella-ähnlicher Erreger steht in enger Verwandtschaft zu einem Erreger, der in modernen afrikanischen Elefanten tödliche Ausbrüche verursacht hat, was die Möglichkeit nahelegt, dass Mammuts eine ähnliche Anfälligkeit für vergleichbare Infektionen geteilt haben könnten.
In einem Meilenstein rekonstruierten die Forschenden Teile eines Erysipelothrix-Genoms aus dem etwa 1,1 Millionen Jahre alten Steppemammut. Dies ist die älteste authentifizierte wirt-assoziierte mikrobielle DNA, die bisher geborgen wurde, und zeigt, dass unter günstigen Bedingungen mikrobielle Genome weit über das Erhaltungsfenster des Wirtsgenoms hinaus überdauern können. Der Fund erweitert den zeitlichen Rahmen für die Untersuchung alter Wirts-Mikroben-Beziehungen und antiker Pathogenökologie.

Scientific context and implications
Wirt-assoziierte Mikroben evolvieren relativ schnell im Vergleich zu ihren großen vielzelligen Wirten; daher bietet die Wiederherstellung ihrer Genome aus tiefen Zeitabschnitten eine Möglichkeit, mikrobiellen Wandel und Wirts-Pathogen-Dynamiken über evolutionäre Zeiträume zu untersuchen. Diese Ergebnisse deuten darauf hin, dass bestimmte mikrobielle Linien über weite geografische Bereiche und lange Zeiträume mit Mammuts koexistierten — von vor mehr als einer Million Jahren bis hin zum späten Überleben der Wollmammuts auf Wrangel Island vor etwa 4.000 Jahren.
Tom van der Valk, Seniorautor der Studie, betont, dass alte Überreste biologische Informationen jenseits des Wirtsgenoms bewahren können und dass diese mikrobiellen Archive neue Perspektiven darauf liefern, wie Mikroben Anpassung, Krankheit und Aussterben in Pleistozän-Ökosystemen beeinflusst haben. Gleichzeitig mahnen die Autorinnen und Autoren zur Vorsicht: DNA-Abbau, verzerrende Zerfallsprozesse und begrenzte Vergleichsdatenbanken erschweren es, die exakte Auswirkung dieser Mikroben auf die Mammutgesundheit mit völliger Sicherheit zu bestimmen.

Limitations and cautions
Studien zur antiken mikrobiellen DNA stehen vor besonderen Herausforderungen: Mikrobielle DNA ist kürzer und kontaminationsanfälliger als Wirbeltier-DNA, moderne Verwandte sind in Referenzdatenbanken oft unterrepräsentiert, und taphonomische Prozesse können Umweltarten in fossiles Material einbringen. Die Forschenden kombinieren mehrere Authentifizierungs-Kriterien, um diese Probleme zu mindern, doch weitere Arbeiten — insbesondere der Ausbau von Referenzgenomen für Wildtier-Mikroben — werden die Interpretationen stärken.
Technologies and future prospects
Fortschritte in Sequenzierungstechnologie, metagenomischer Assemblierung und maschinellem Lernen waren entscheidend für diese Studie. Mit wachsenden Referenzdatenbanken für mikrobielle Genome und verbesserten Rechenmethoden wird die Paläomikrobiologie besser in der Lage sein, alte mikrobielle Gemeinschaften zu rekonstruieren, Evolutionsraten abzuschätzen und Gene zu identifizieren, die mit Virulenz oder Wirtspezifität assoziiert sind. Ethische und praktische Überlegungen werden jede Bemühung begleiten, antike Mikroben zu synthetisieren oder funktional zu charakterisieren; der primäre Wert dieser Befunde liegt im Verständnis von Evolution, Ökologie und Krankheitsdynamik und nicht in der Wiederbelebung ausgestorbener Arten.
Applications beyond paleontology
Die Rekonstruktion antiker Mikroben liefert Erkenntnisse für die heutige Naturschutzbiologie, indem sie historische Baselines für wirt-assoziierte Mikrobiome klärt und die Evolution von Krankheitserregern in nahen Verwandten ausgestorbener Arten wie afrikanischen und asiatischen Elefanten beleuchtet. Einsichten in vergangene Wirts-Pathogen-Interaktionen können Veterinäre und Wildtiergesundheitsexperten dabei unterstützen, langfristige Krankheitsdynamiken zu bewerten und Risiken für gefährdete Arten abzuschätzen.
Expert Insight
Dr. Elena Rossi, eine fiktive Evolutionsmikrobiologin am Institute for Ancient Genomics, kommentiert: "Die Gewinnung mikrobieller DNA, die über eine Million Jahre alt ist, ist ein technischer und konzeptioneller Meilenstein. Sie verändert die Fragen, die wir zur Koevolution stellen können — von punktuellen Momentaufnahmen hin zu Zeitreihen, die zeigen, wie sich mikrobielle Gemeinschaften veränderten, während Wirte sich an wandelnde Klimata und Landschaften anpassten. Diese Arbeit unterstreicht auch die Notwendigkeit umfangreicherer mikrobieller Referenzdatenbanken, um alte Signale präziser zu interpretieren."
Future directions
Zu den nächsten Schritten gehören gezielte Anreicherungen potenzieller Pathogen-Genome, breitere geografische Probenahmen von Megafauna-Überresten und vergleichende Studien mit lebenden Elefanten und anderen Proboscidea. Forschende werden zudem verbesserte Methoden zur Authentifizierung und Assemblierung degradierter mikrobieller Genome verfolgen und versuchen, spezifische mikrobiellen Gene mit Merkmalen wie Antibiotikaresistenz oder Virulenz zu verknüpfen. Interdisziplinäre Kooperationen — die Paläogenomik, Mikrobiologie, Ökologie und Veterinärwissenschaften vereinen — werden entscheidend sein, um das volle Potenzial der Forschung am Mikrobiom der Tiefzeit auszuschöpfen.
Conclusion
Die Gewinnung mikrobieller DNA aus Mammut-Überresten, die mehr als eine Million Jahre alt sind, zeigt, dass fossile Materialien Spuren der Mikroben bewahren können, die mit ausgestorbenen Tieren lebten. Die Identifizierung persistenter mikrobieller Kladen — einschließlich potenzieller Pathogene — bietet ein einzigartiges Fenster auf die Koevolution von Wirten und Mikroben im Pleistozän und eröffnet neue Wege zur Untersuchung von Krankheit, Anpassung und Aussterben. Trotz technischer und interpretativer Herausforderungen markiert diese Studie einen bedeutenden Fortschritt für die Paläomikrobiologie und erweitert unser Verständnis davon, wie Mikroben die Geschichte des Lebens auf der Erde geprägt haben.
Quelle: scitechdaily

Kommentare